PCR: Arten und Anwendungen
PCR: Arten und Anwendungen
Die PCR ist ein Eckpfeiler der Molekularbiologie und ermöglicht es Wissenschaftlern, DNA-Fragmente mit unglaublicher Präzision zu vergrößern und zu analysieren. In diesem Blog befassen wir uns mit dem Verständnis der PCR – von ihren Grundprinzipien bis hin zu den verschiedenen Techniken, die Forschung, Diagnostik und darüber hinaus revolutioniert haben.
Wichtige Erkenntnisse
1. PCR ist eine zentrale Technik zur DNA-Amplifikation, die in der Forschung, Diagnostik und Forensik von entscheidender Bedeutung ist.
2. Varianten wie Hot Start PCR, RT-PCR und qPCR verbessern die Spezifität, analysieren RNA und quantifizieren Nukleinsäuren.
3. Fortschritte wie High-Fidelity-Polymerase und digitale PCR bieten Präzision beim Nachweis von Mutationen und Genen mit geringer Häufigkeit.
Inhaltsverzeichnis
Zu einem Abschnitt springen:
PCR
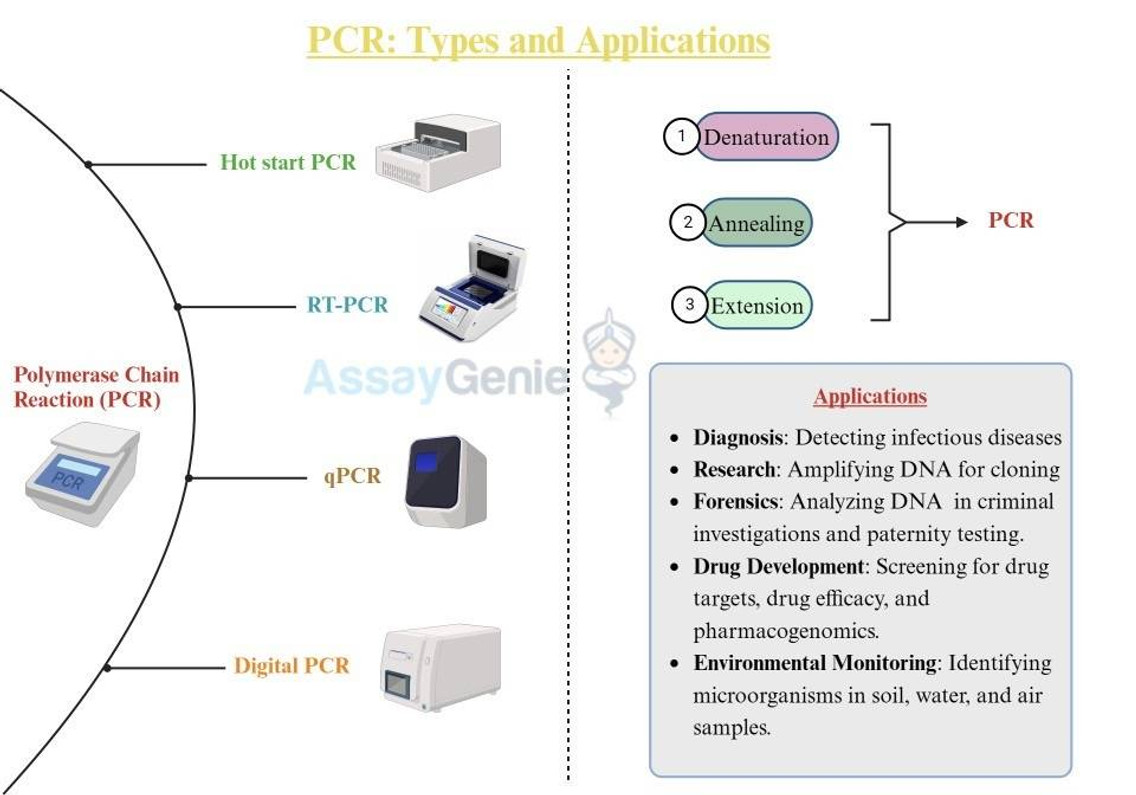
Die Polymerase-Kettenreaktion (PCR) ist eine leistungsstarke Labortechnik, mit der bestimmte DNA-Segmente amplifiziert werden, wodurch Millionen von Kopien aus einem einzigen DNA-Stück erstellt werden. Die in den 1980er Jahren entwickelte PCR hat sich zu einem unverzichtbaren Werkzeug in verschiedenen Bereichen der biologischen Forschung, Diagnostik, Forensik und mehr entwickelt. Es revolutionierte die DNA-Analyse, indem es Wissenschaftlern ermöglichte, selbst aus begrenzten oder degradierten Proben ausreichende Mengen an DNA für die Analyse herzustellen.
Der PCR-Prozess umfasst eine Reihe temperaturabhängiger Schritte und nutzt ein DNA-Polymerase-Enzym, um die Synthese neuer DNA-Stränge zu katalysieren. Hier finden Sie eine Schritt-für-Schritt-Erklärung des PCR-Prozesses:
1. Denaturierung (94–98 °C): Die doppelsträngige DNA-Matrize wird auf eine hohe Temperatur erhitzt, normalerweise etwa 94–98 °C. Dadurch werden die Wasserstoffbrückenbindungen zwischen den komplementären DNA-Strängen aufgebrochen, was zur Trennung der beiden Stränge führt. Dieser Schritt wird oft als „Schmelzen“ bezeichnet.
2. Annealing (50–65 °C): Die Temperatur wird auf einen bestimmten Bereich (typischerweise 50–65 °C) gesenkt, wodurch kurze DNA-Primer an die komplementären Sequenzen am Anfang der Ziel-DNA-Region binden können. Diese Primer dienen als Ausgangspunkte für die DNA-Synthese.
3. Verlängerung (72 °C): Die Temperatur wird auf etwa 72 °C erhöht. Dies ist die optimale Temperatur für das DNA-Polymerase-Enzym (normalerweise Taq-Polymerase), um einen neuen DNA-Strang zu synthetisieren, indem zum Matrizenstrang komplementäre Nukleotide hinzugefügt werden. Die Primer bestimmen die Sequenz des neu synthetisierten DNA-Strangs.
Diese drei Schritte – Denaturierung, Annealing und Verlängerung – werden typischerweise für eine bestimmte Anzahl von Zyklen wiederholt, normalerweise etwa 20 bis 40 Zyklen. Jeder Zyklus verdoppelt die DNA-Menge, was zu einem exponentiellen Anstieg des Ziel-DNA-Segments führt.

Abb: Polymerase-Kettenreaktion (PCR)
Standard-PCR-Techniken stoßen beim Umgang mit Guanin-/Cytosin-reichen (GC-reichen) Bereichen auf eine Hürde. Sequenzen mit mehr GC sind stabiler als solche mit weniger GC. Darüber hinaus erzeugen GC-reiche Sequenzen eher Sekundärstrukturen wie Haarnadelschleifen. Daher ist es während des Denaturierungsschritts schwierig, GC-reiche Doppelstränge vollständig zu trennen. Infolgedessen stößt die DNA-Polymerase auf Schwierigkeiten bei der Bildung des neuen Strangs. Dies kann durch Änderungen zu einer höheren Denaturierungstemperatur und kürzeren Annealing-Zeiten verbessert werden, die auch dazu beitragen, die unspezifische Bindung von GC-reichen Primern zu verhindern. Die Amplifikation GC-reicher Sequenzen kann durch Zugabe zusätzlicher Reagenzien verbessert werden. Aufgrund der durch GC-Wechselwirkungen hervorgerufenen Störung der Sekundärstrukturen wird die Trennung der Doppelstränge durch die Anwendung von DMSO, Glycerin und Betain erleichtert.
Hot-Start-PCR
Die Hot-Start-PCR ist eine modifizierte Version der traditionellen Polymerase-Kettenreaktionstechnik (PCR), die darauf ausgelegt ist, die Spezifität und Effizienz der DNA-Amplifikation zu verbessern. Es behebt ein häufiges Problem bei der Standard-PCR, bei dem es aufgrund der vorzeitigen Aktivierung des DNA-Polymerase-Enzyms in der Anfangsphase des Reaktionsaufbaus zu unspezifischer Amplifikation kommen kann. Dies kann zur Bildung unerwünschter Produkte und einer verringerten Gesamtspezifität führen. Bei der Hot-Start-PCR werden Maßnahmen ergriffen, um zu verhindern, dass die DNA-Polymerase bei niedrigen Temperaturen aktiv wird, beispielsweise während der Ersteinrichtung und den ersten Erhitzungsschritten des PCR-Prozesses. Ziel ist es, die Amplifikation unspezifischer Produkte zu verhindern und die Amplifikation der beabsichtigten Zielsequenz zu verbessern. Dies ist besonders wertvoll, wenn mit komplexen DNA-Vorlagen gearbeitet wird oder wenn es um Proben geht, die Spuren von Ziel-DNA enthalten. Zu den Vorteilen der Hot-Start-PCR gehören eine verbesserte Spezifität, eine erhöhte Empfindlichkeit und eine geringere Optimierung.
Es gibt verschiedene Methoden zur Implementierung der Hot-Start-PCR-Technik:
Physische Trennung: Ein gängiger Ansatz besteht darin, die DNA-Polymerase während der Ersteinrichtung physisch von ihrer DNA-Matrize und ihren Primern zu trennen. Diese Trennung wird durch den Einsatz modifizierter DNA-Polymerasen oder antikörperbasierter Techniken erreicht. Beispielsweise kann ein Antikörper verwendet werden, um die Aktivität der Polymerase zu hemmen, bis die Reaktion erhitzt wird. Zu diesem Zeitpunkt denaturiert der Antikörper und ermöglicht es der Polymerase, aktiv zu werden.
Chemische Modifikation: Eine andere Methode beinhaltet die chemische Modifikation des DNA-Polymerase-Enzyms selbst. Dem Enzym werden chemische Modifikationen hinzugefügt, die es bei niedrigeren Temperaturen inaktiv machen. Wenn die Reaktionstemperatur während der ersten Erhitzungsschritte steigt, werden diese Modifikationen entfernt und das Enzym für die DNA-Amplifikation aktiviert.
Hot-Start-Taq-Polymerase: Einige kommerzielle DNA-Polymerasen sind so konzipiert, dass sie bei Raumtemperatur oder niedriger inaktiv sind, bei erhöhten Temperaturen jedoch aktiv werden. Diese spezialisierten Enzyme, die oft als „Hot-Start-Taq-Polymerasen“ bezeichnet werden, verfügen über eine durch Antikörper vermittelte Hemmung, reversible chemische Modifikationen oder andere Mechanismen, um ihre Aktivität zu verhindern, bis die Reaktion richtig erhitzt ist.
High-Fidelity-Polymerase
Fehler beim Nukleotidabgleich können auftreten, obwohl DNA-Polymerasen recht präzise auf die ursprüngliche Matrizensequenz amplifizieren. Bei Anwendungen wie dem Klonen können Fehlpaarungen zu verkürzten Transkripten und Proteinen führen, die falsch übersetzt wurden oder inaktiv sind. Es wurden Polymerasen mit einer „Korrekturlese“-Aktivität gefunden und in den Arbeitsablauf integriert, um diese Nichtübereinstimmungen zu verhindern. In Pyrococcus furiosus wurde 1991 die erste korrekturlesende Polymerase, Pfu, entdeckt. Die 3'-zu-5'-Exonukleaseaktivität dieses Pfu-Enzyms. Die Exonuklease eliminiert fehlgepaarte Nukleotide am 3'-Ende des Strangs, während die DNA amplifiziert wird. Dann wird das richtige Nukleotid ersetzt und die DNA-Synthese geht weiter.
Die richtige Bindungsaffinität des Nukleosidtriphosphats zum Enzym, bei der eine ineffektive Bindung die Synthese verzögert und den richtigen Ersatz ermöglicht, wird zur Identifizierung fehlerhafter Nukleotidsequenzen verwendet. Im Vergleich zur Taq-DNA-Polymerase führt die Korrekturleseaktivität der Pfu-Polymerase zu weniger Fehlern in der endgültigen Sequenz. Um die Fehlerquote bei der DNA-Amplifikation weiter zu senken, wurden kürzlich zusätzliche Korrekturleseenzyme entdeckt und das ursprüngliche Pfu-Enzym modifiziert.
RT-PCR
Die Reverse-Transkription-Polymerase-Kettenreaktion, allgemein bekannt als RT-PCR, ist eine leistungsstarke molekularbiologische Technik, die die Prinzipien der Reverse-Transkription und der Polymerase-Kettenreaktion kombiniert. Diese Methode ist speziell darauf ausgelegt, RNA-Moleküle zu analysieren und zu verstärken und sie zur weiteren Analyse in komplementäre DNA (cDNA) umzuwandeln.
Der Prozess beginnt mit dem Schritt der reversen Transkription, bei dem ein Enzym namens Reverse Transkriptase einen komplementären DNA-Strang (cDNA) unter Verwendung eines einzelsträngigen RNA-Moleküls als Vorlage synthetisiert. Dieser Schritt ist von entscheidender Bedeutung, da an vielen biologischen Prozessen RNA beteiligt ist, beispielsweise an der Genexpression und der Virusreplikation. Durch die Umwandlung von RNA in cDNA können Forscher diese Prozesse einfacher untersuchen.
Sobald die cDNA erzeugt ist, wird die Polymerasekettenreaktion eingesetzt, um spezifische Zielsequenzen zu amplifizieren. Dies beinhaltet wiederholte Zyklen des Erhitzens und Abkühlens der Reaktionsmischung. Während des Erhitzungsschritts (Denaturierung) werden die DNA-Stränge getrennt, wodurch einzelsträngige Matrizen entstehen. Im Abkühlschritt (Annealing) heften sich kurze DNA-Primer, die speziell für die Bindung an die Ziel-cDNA-Sequenzen entwickelt wurden, an die Templates. Anschließend verlängert ein hitzestabiles DNA-Polymerase-Enzym die Primer durch Hinzufügen komplementärer Nukleotide, was zur Synthese neuer DNA-Stränge führt.
Das Endergebnis der RT-PCR ist eine amplifizierte Menge an cDNA aus der ursprünglichen RNA-Probe, die es Forschern ermöglicht, die Genexpressionsniveaus zu untersuchen, Virusinfektionen zu erkennen, RNA-Sequenzen zu analysieren und vieles mehr. RT-PCR hat sich in verschiedenen Bereichen als unschätzbar wertvoll erwiesen, von der medizinischen Diagnostik, wo sie zum Nachweis von Krankheiten wie COVID-19 eingesetzt wird, bis hin zur molekularbiologischen Forschung, wo sie die Erforschung der Genfunktion und -regulation ermöglicht. Ihre Fähigkeit, RNA in DNA umzuwandeln und dann spezifische Sequenzen zu amplifizieren, hat RT-PCR zu einem unverzichtbaren Werkzeug in der modernen Molekularbiologie gemacht.
qPCR und RT-qPCR
Für viele Anwendungen werden Nukleinsäuren mittels quantitativer PCR (qPCR) nachgewiesen, beschrieben und quantifiziert. Bei der RT-qPCR werden RNA-Transkripte häufig quantifiziert, indem, wie bereits erwähnt, zunächst in cDNA revers transkribiert und anschließend eine qPCR durchgeführt wird. Denaturierung, Annealing und Elongation sind drei Prozesse, die zur Amplifikation der DNA wiederholt werden, ähnlich wie bei der herkömmlichen PCR. Bei der qPCR ermöglicht die Fluoreszenzmarkierung jedoch die Erfassung von Daten im Verlauf der PCR. Aufgrund der Vielfalt der verfügbaren Techniken und Chemikalien bietet diese Technologie mehrere Vorteile.
Die Fluoreszenzmarkierung, die einen dsDNA-bindenden Farbstoff verwendet, ermöglicht die Messung der amplifizierten DNA-Moleküle in der farbstoffbasierten qPCR (normalerweise grün). Die Fluoreszenz wird über jeden Zyklus hinweg gemessen. Die Menge der verdoppelten DNA führt zu einem entsprechenden Anstieg des Fluoreszenzsignals. Dadurch wird die DNA „in Echtzeit“ gemessen. Mit der farbstoffbasierten qPCR kann jeweils nur ein Ziel analysiert werden, und jede in der Probe gefundene ds-DNA führt zur Bindung des Farbstoffs.
Mit der sondenbasierten qPCR können in jeder Probe viele Ziele gleichzeitig identifiziert werden. Dazu ist jedoch die Erstellung und Entwicklung einer oder mehrerer zielspezifischer Sonden erforderlich, die zusätzlich zu den Primern eingesetzt werden. Es gibt viele andere Arten von Sondendesigns, aber die beliebteste Art ist eine Hydrolysesonde, die einen Fluorophor mit einem Quencher kombiniert. Wenn die Sonde noch intakt ist, stoppt der Fluoreszenzresonanzenergietransfer (FRET) die Emission des Fluorophors durch den Quencher. Allerdings wird die Sonde während der Primerverlängerung und Amplifikation der jeweiligen Sequenz, mit der sie während der PCR-Reaktion verknüpft ist, hydrolysiert. Durch die Spaltung der Sonde kommt es zu einem verstärkungsabhängigen Anstieg der Fluoreszenz, wodurch der Fluorophor vom Quencher befreit wird.
Dadurch korreliert die Menge der in der Probe vorhandenen Sondenzielsequenz direkt mit dem Fluoreszenzsignal eines sondenbasierten qPCR-Prozesses. In qPCR-basierten diagnostischen Tests wird häufig die sondenbasierte qPCR eingesetzt, da sie präziser ist als die farbstoffbasierte qPCR.

Abb: Schritte in qPCR
Isotherme Verstärkung
Damit die oben diskutierten PCR-Methoden die Kammertemperaturen für die Denaturierungs-, Annealing- und Verlängerungsphasen präzise erhöhen und senken können, sind teure Thermocycling-Geräte erforderlich. Es wurden zahlreiche Methoden entwickelt, die keine derart genaue Ausrüstung erfordern und innerhalb der Zielzellen oder sogar in einem einfachen Wasserbad verwendet werden können. Diese Methoden, die zusammenfassend als isotherme Verstärkung bezeichnet werden, basieren auf den Prinzipien der exponentiellen, linearen oder kaskadenartigen Verstärkung.
Die schleifenvermittelte isotherme Verstärkung (LAMP) ist die bekannteste Art der isothermen Verstärkung. LAMP amplifiziert Template-DNA oder -RNA mittels exponentieller Amplifikation bei 650 °C. Mithilfe einer DNA-Polymerase und vier bis sechs Primern, die zu bestimmten Abschnitten der Ziel-DNA komplementär sind, erzeugt LAMP neue DNA. In der frisch synthetisierten DNA könnte sich eine „Schleifen“-Struktur bilden, wenn die komplementären Sequenzen zweier dieser Primer Sequenzen in den anderen Primern erkennen und an sie binden. Diese Struktur erleichtert dann das Primer-Annealing in nachfolgenden Amplifikationsrunden. Zur Darstellung von LAMP können zahlreiche Techniken wie Fluoreszenz, Agarosegelelektrophorese oder Kolorimetrie eingesetzt werden.
LAMP war eine geeignete Alternative für SARS-CoV-2-Tests an Orten, an denen klinische Labortests nicht leicht zugänglich waren, wo Probenlagerung und -transport nicht praktikabel waren, oder in Laboren, die bisher nicht über PCR-Thermocycling-Geräte verfügten, weil diese einfach zu visualisieren und zu testen waren Das Vorhandensein oder Nichtvorhandensein eines Produkts lässt sich kolorimetrisch nachweisen und erforderte keine teure Ausrüstung.
Digitale PCR
Die digitale Polymerase-Kettenreaktion oder digitale PCR ist eine hochmoderne molekularbiologische Technik, die die Prinzipien der traditionellen PCR auf ein neues Maß an Präzision und Empfindlichkeit hebt. Mithilfe der digitalen PCR können in einer Probe vorhandene DNA- oder RNA-Moleküle genau quantifiziert und analysiert werden, selbst wenn sie in sehr geringen Konzentrationen vorhanden sind.
Bei der digitalen PCR wird die Probe in Tausende von Einzelreaktionen aufgeteilt, von denen jede ein einzelnes Molekül oder einige Moleküle der Ziel-DNA oder -RNA enthält. Diese Aufteilung wird mithilfe mikrofluidischer Geräte oder spezieller Emulsionstechniken erreicht. Jede geteilte Reaktion fungiert als Miniatur-PCR-Reaktion, wobei die Ziel-DNA/RNA je nach Vorhandensein oder Fehlen entweder amplifiziert wird oder nicht amplifiziert bleibt.
Nach der Amplifikation werden die Partitionen analysiert und die Anzahl der positiven Partitionen (diejenigen, bei denen eine Amplifikation stattgefunden hat) und der negativen Partitionen (diejenigen ohne Amplifikation) gezählt. Anhand dieser Informationen wird die absolute Menge der Zielmoleküle in der Originalprobe berechnet. Die Fähigkeit der digitalen PCR, seltene Sequenzen mit hoher Präzision zu erkennen und zu quantifizieren, macht sie besonders wertvoll für Anwendungen wie die Erkennung genetischer Mutationen, die Untersuchung der Genexpression auf niedrigem Niveau und die Analyse komplexer DNA- oder RNA-Mischungen.
Im Vergleich zur herkömmlichen quantitativen PCR (qPCR) bietet die digitale PCR mehrere Vorteile, darunter eine höhere Empfindlichkeit, eine geringere Anfälligkeit für PCR-Inhibitoren und eine verbesserte Genauigkeit für Ziele mit geringer Häufigkeit. Es ist ein leistungsstarkes Werkzeug in verschiedenen Bereichen, darunter medizinische Diagnostik, Gentests, Umweltüberwachung und mehr, wo eine genaue Quantifizierung von Nukleinsäuren von entscheidender Bedeutung ist. Durch die Bereitstellung einer digitalen, absoluten Quantifizierung von Zielmolekülen trägt die digitale PCR zu zuverlässigeren und präziseren molekularen Analysen bei.
Zusammenfassend unterstreicht diese Untersuchung der Polymerasekettenreaktion (PCR) und ihrer verschiedenen Arten die überragende Bedeutung dieser revolutionären molekularbiologischen Technik. Durch die geschickte Amplifikation und Analyse von Nukleinsäuren ermöglichen uns PCR-Techniken, komplizierte genetische Informationen zu entschlüsseln und bisher unzugängliche Erkenntnisse zu entschlüsseln. Unsere Untersuchung hat sich mit den Feinheiten von PCR-Varianten befasst und umfasst die Reverse Transkriptions-PCR (RT-PCR) und ihre vielfältigen Anwendungen. Die Erläuterung dieser Methoden und ihrer inhärenten Fähigkeiten unterstreicht die entscheidende Rolle, die sie in der modernen molekularbiologischen Forschung und Diagnostik spielen
3rd Sep 2024
Recent Posts
-
Neuroimmunologie: Das Immunsystem des ZNS
…30th Sep 2024 -
Interleukin-8-Signalisierung
IL-8-ÜbersichtInterleukin-8 (IL-8), auch bekannt als CXCL8, wurde vor über 30 Jahren a …25th Sep 2024 -
Waardenburg-Syndrom und Klein-Waardenburg-Syndrom
Waardenburg-Syndrom: Eine seltene genetische Störung, die Pigmentierung, Hörvermögen u …25th Sep 2024